
单细胞蛋白组旨在通过高分辨率的质谱技术对单个细胞内的蛋白质进行全面解析。这一技术突破了传统整体蛋白质组学的局限性,能够深入揭示细胞间的异质性及其在复杂生物系统中的作用。每个细胞都是一个独立的微型生物工厂,内部蛋白质的种类、含量及修饰状态直接决定了细胞的功能与状态。然而,由于细胞间的高度异质性,即使在同一个组织中,细胞之间的功能和响应机制也可能存在显著差异。传统的蛋白质组学方法往往只能提供整体平均信息,无法揭示单个细胞层面的独特特征。而单细胞蛋白组技术能够将组织解离成单个细胞,并对每个细胞进行独立的蛋白组分析,从而揭示细胞间的差异性及其生物学意义。这对于肿瘤微环境、免疫细胞功能、发育生物学及药物响应机制等领域具有重要的应用价值。例如,在肿瘤研究中,不同细胞亚群的特定蛋白表达谱往往与肿瘤的侵袭性、转移能力和治疗耐药性密切相关。通过单细胞蛋白组分析,我们不仅可以鉴定关键的蛋白分子,还能够构建细胞间相互作用网络,揭示细胞行为背后的分子机制。此外,单细胞蛋白组还能够捕捉到细胞在不同时间点的动态变化,为揭示细胞分化、发育和疾病进展的过程提供强有力的工具。百泰派克生物科技使用全流程集成自动化的单细胞分选平台cellenONE分选得到的单细胞使用基本无损的蛋白前处理方法酶切,得到的肽段使用最新的,高灵敏度的Thermo Orbitrap Astral设备上机检测,保证数据质量。
一、项目概述
1、项目信息

表1. 项目信息表

表2. 样本信息表
注:实验室收到样本后对样本进行编号,后均以样本编号代替样本名称。
二、实验说明
1、实验流程总览
本实验对客户提供的样品进行单细胞蛋白组分析,主要实验流程如下:

图1
2、实验材料与设备
(1)试剂耗材
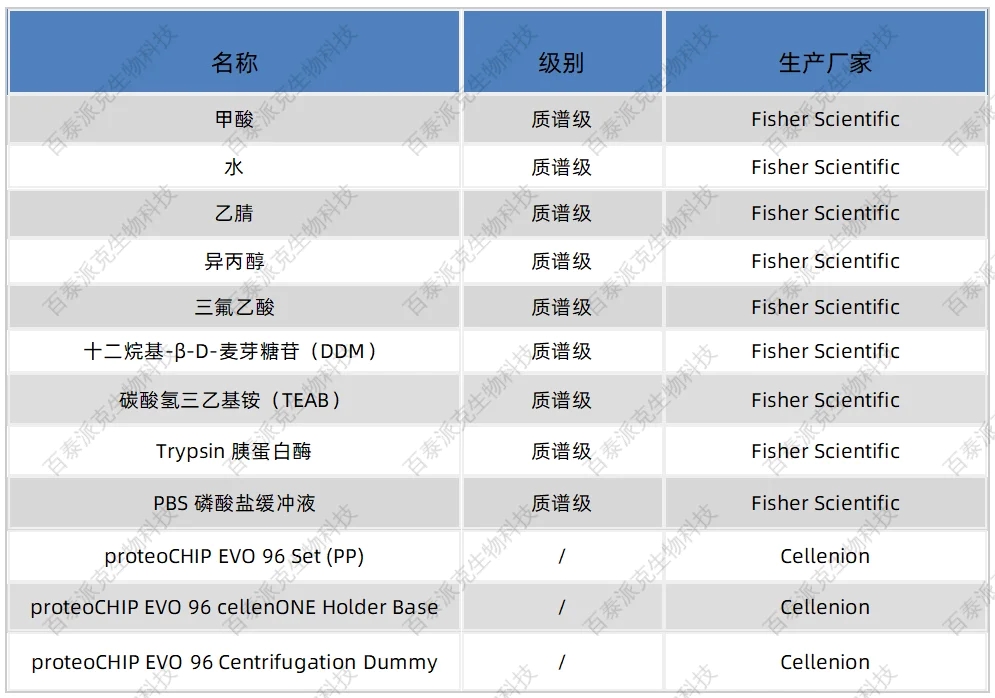
表3
(2)仪器设备

表4
3、实验方法
(1)样品处理
① 收集细胞:100~200 细胞 /µL。
② 铺板:使用cellenOne 每孔加入 1µL 的 master mix buffer。
③ Mapping:cellenOne 自动进样针吸取 5~10µL 细胞溶液,进行细胞Mapping,判断细胞形态、密度、直径是否符合要求,获取 Mapping 报告。
④ 分选:根据Mapping结果,设置细胞直径范围和Elongation factor(通常为 1.5~1.8),进行细胞分选,最后获取分选结果。
⑤ 补液:细胞分选完成后,使用cellenOne每孔加入 500nL master mix buffer。
⑥ 酶切:设置cellenOne样品盘温度为50°C,湿度85%,恒温恒湿下,进行酶切反应1h,并开启全程自动循环补水500nL/孔,反应结束后,样品盘温度冷却至 20°C。
⑦ 淬灭:每孔手动加入 3.5µL 0.1%TFA/1% DMSO。
⑧ 封板和上样:96/384 孔板盖子封板后置于 Vanquish Neo 中,上样 4µL,进行数据采集。
(2)LC-MS/MS检测
① 液相色谱条件
● 流动相A:0.1% 甲酸水。
● 流动相B:80% 乙腈 /0.1% 甲酸水。
● 流速:0.2μL/min。
● 色谱柱:Ionopticks Aurora Elite 15cm x 75 μm ID, 1.7 μm C18 (包含 emitter),柱温60℃。
● AS样品盘温度:7°C。
● 洗针液:强洗:80%乙腈/0.1%甲酸水;弱洗:0.1%甲酸水。
● 洗针模式:After Draw。
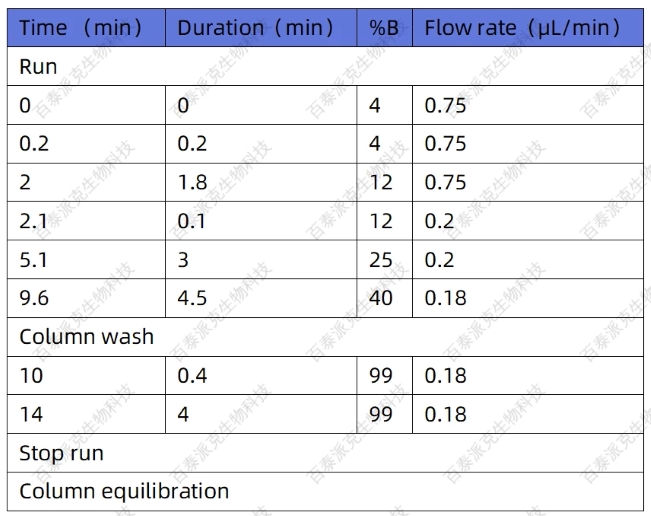
表5
② 质谱条件
● 一级质谱参数:
Resolution:240,000
AGCtarget:500%
MaximumIT:100 ms
Scanrange:400 to 800m/z
● 二级质谱参数:
Scanrange:150 to 2000m/z
AGCtarget:800%
三、数据分析
1、数据分析流程总览
基于质谱下机数据,使用数据库搜索软件进行肽段和蛋白质的鉴定和定量;通过肽段序列长度分布等分析来评估质谱检测数据的质量;采用降维分群方法对单细胞进行分析,结合差异统计,分析各分群差异蛋白;最后对差异蛋白进行GO、KEGG功能富集等一系列功能分析,探索蛋白在单细胞维度的异质性表达和功能特征。生物信息学分析总体流程如下图:
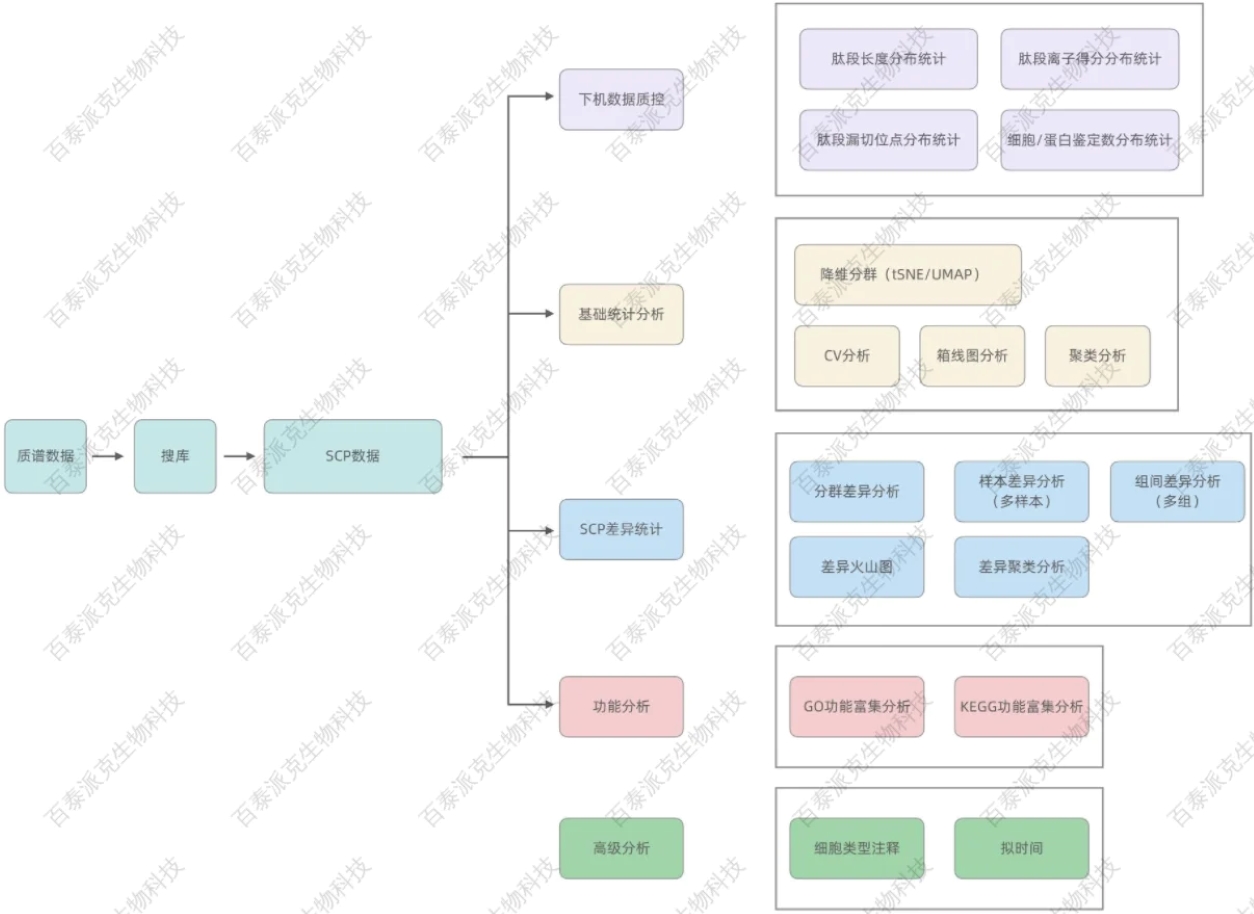
图2
2、数据库检索
质谱原始文件使用DIANN(version) 分别检索目标蛋白数据库,检索参数如下:
(1)固定修饰(Fixed modifications):Carbamidomethyl (C)
(2)可变修饰(Variable modifications):Acetyl (N-term), Oxidation (M)
(3)酶(Enzyme):Trypsin
(4)数据库(Database):uniprot_Homo sapiens (Human)
(5)遗漏酶切位点(Maximum Missed Cleavages):3
(6) 一级质谱误差(Peptide Mass Tolerance):20 ppm
(7) 二级质谱误差(Fragment Mass Tolerance):0.02 Da
3、鉴定结果总览
基于数据库检索结果,我们统计各细胞蛋白/肽段鉴定数目,最终鉴定结果如下:
表6. 鉴定结果统计表
注:(1)Protein Counts:蛋白鉴定数;(2)Peptide Counts:肽段鉴定数。
4、数据质控
基于数据库检索结果,我们统计肽段长度分布和肽段离子得分分布,对质谱下机数据进行数据质控。
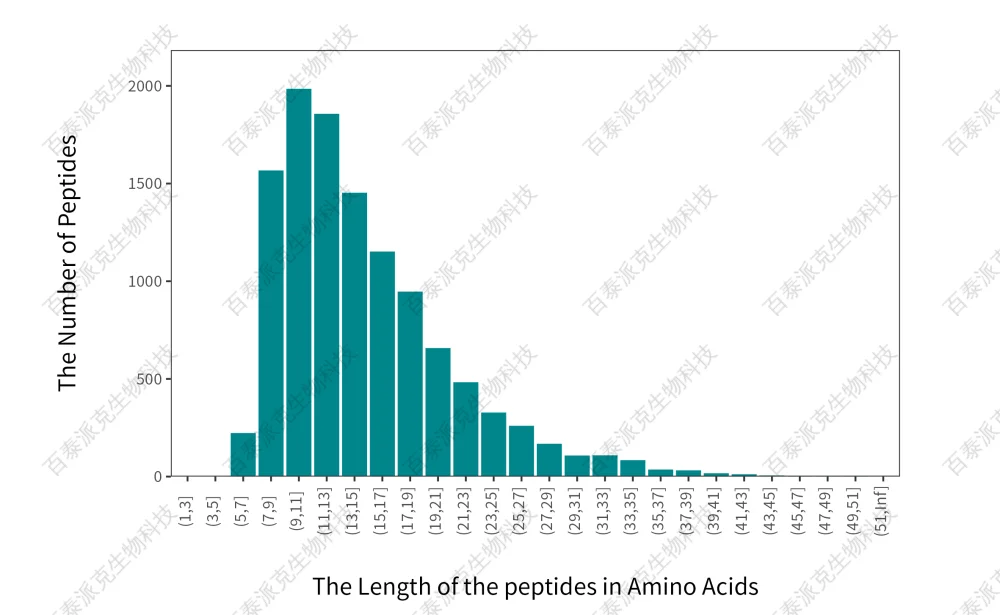
图3. 肽段长度分布图
注:横坐标表示肽段长度分布区间,纵坐标表示区间内肽段数。
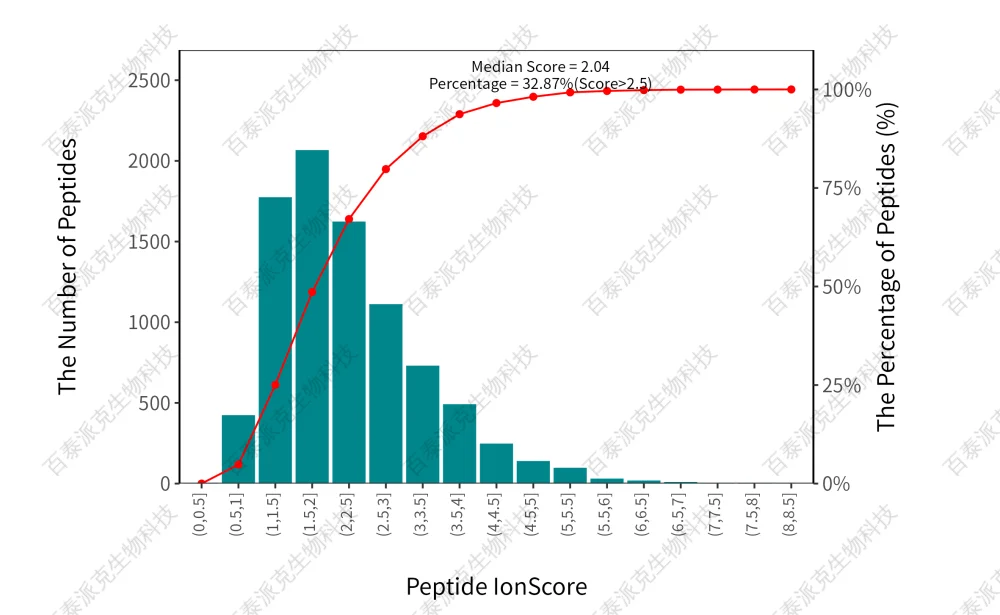
图4. 肽段离子得分分布图
注:横坐标表示肽段离子得分区间,左侧纵坐标表示区间内肽段数,右侧纵坐标表示累计肽段数分布百分比。
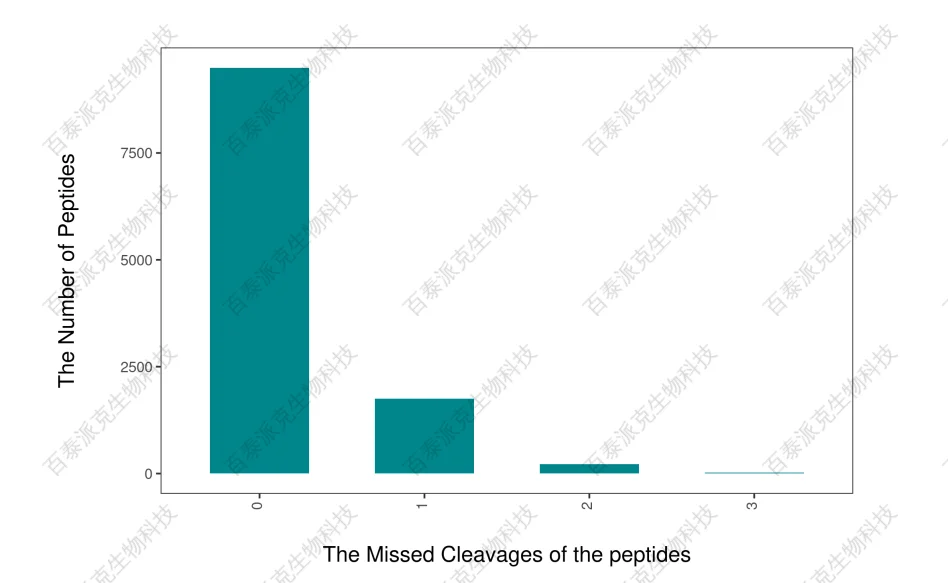
图5. 肽段漏切位点分布图
注:横坐标表示肽段漏切位点数,左侧纵坐标表示不同漏切位点对应的肽段数。
5、降维分群
(1)降维分群
降维指采用某种映射方法,将原高维空间中的数据点映射到低维度的空间中。
PCA是最常用的无监督线性降维方法,能将多个指标转换为少数几个主成分,这些主成分是原始变量的线性组合,且彼此之间互不相关,其能反映出原始数据的大部分信;tSNE和UMAP则属于非线性降维算法,也是目前单细胞数据常用的降维可视化方法。
单细胞聚类分群是基于降维之后的数据根据聚类算法对细胞进行分群聚类,并结合tSNE/UMAP降维结果进行展示。
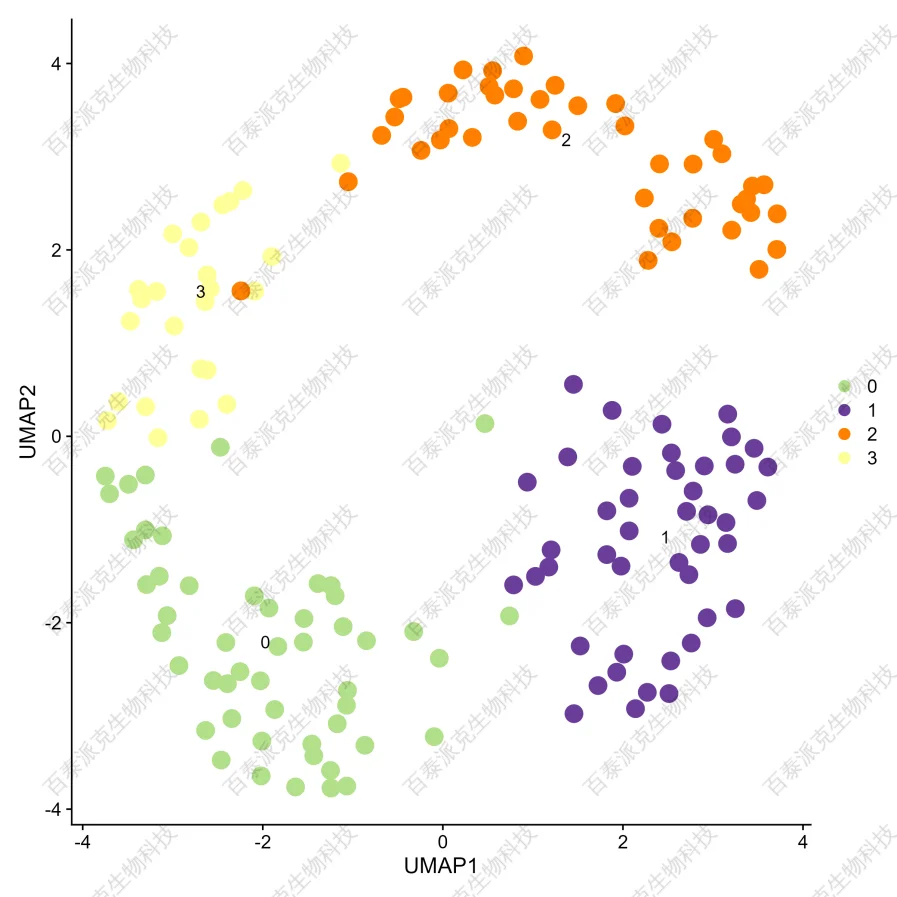
图6
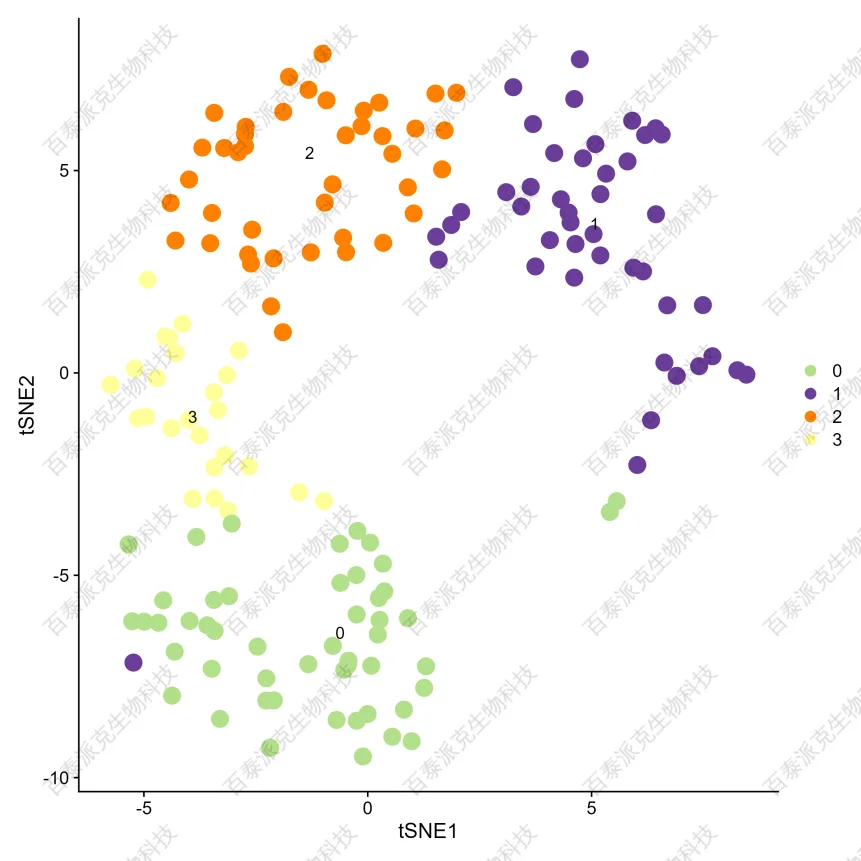
图7. 单细胞降维分群结果(UMAP/tSNE)
注:以不同颜色标注不同的细胞分群结果。
(2)CV分析
在统计学中,变异系数(CV)是标准差与平均值之比,用于描述数据的变异程度。CV值越小,表示数据越稳定,变异程度越小。统计各Cluster内蛋白定量值CV,并以箱线图方式进行展示,可以有效评估Cluster内蛋白是否波动,Cluster间是否差异。
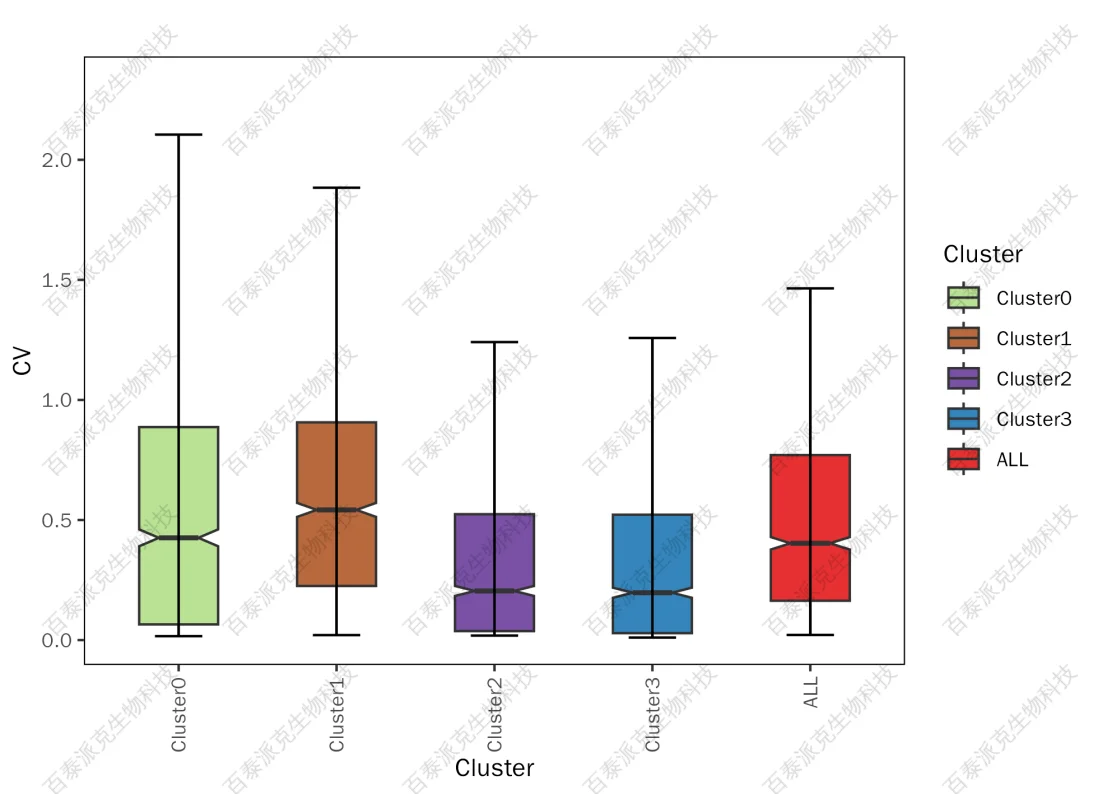
图8. 各Cluster蛋白丰度CV箱线图
注:横坐标表示Cluster,纵坐标表示Cluster内定量值CV的分布区间。
(3)Cluster数据分布
箱线图是对数据分布的一种常用表示方法,因形状如箱子而得名。它利用上四分位数、中位数、下四分位数等几个统计量可以粗略观察数据是否对称,分布的离散程度等。
结合分群Cluster结果,统计Cluster内蛋白在各细胞中平均丰度水平。再以箱线图形式对平均丰度情况进行可视化,可以对不同Cluster内蛋白平均情况进行整体观察比较。
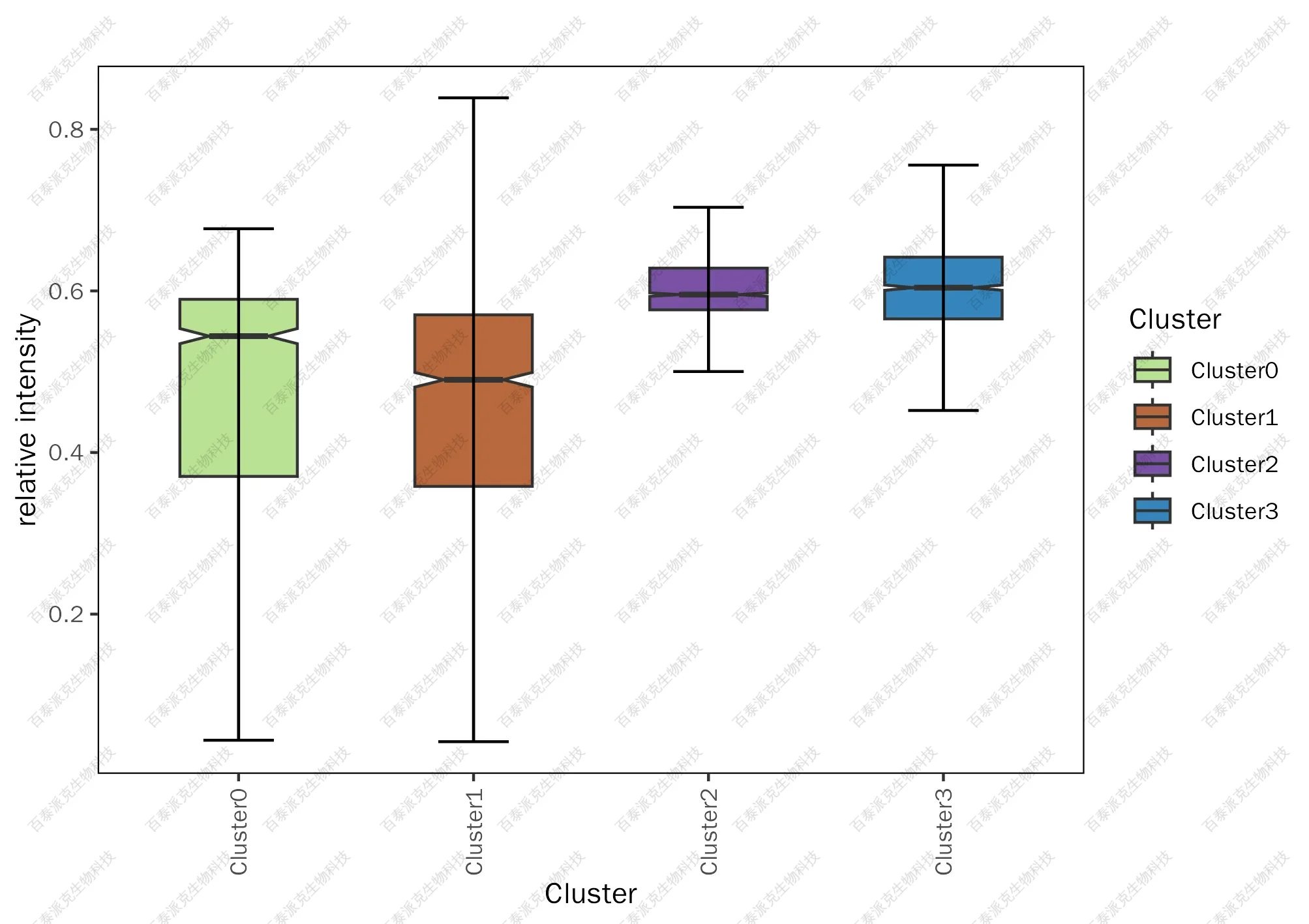
图9. 各Cluster蛋白平均丰度箱线图
注:横坐标表示样本,纵坐标表示定量值分布区间。
(4)聚类热图
基于蛋白在不同细胞内的丰度水平,结合降维分群的信息,以聚类热图的形式进行展示,可以对所有Cluster内各细胞蛋白丰度的趋势进行观察,也可以对不同Cluster蛋白丰度趋势进行比较。
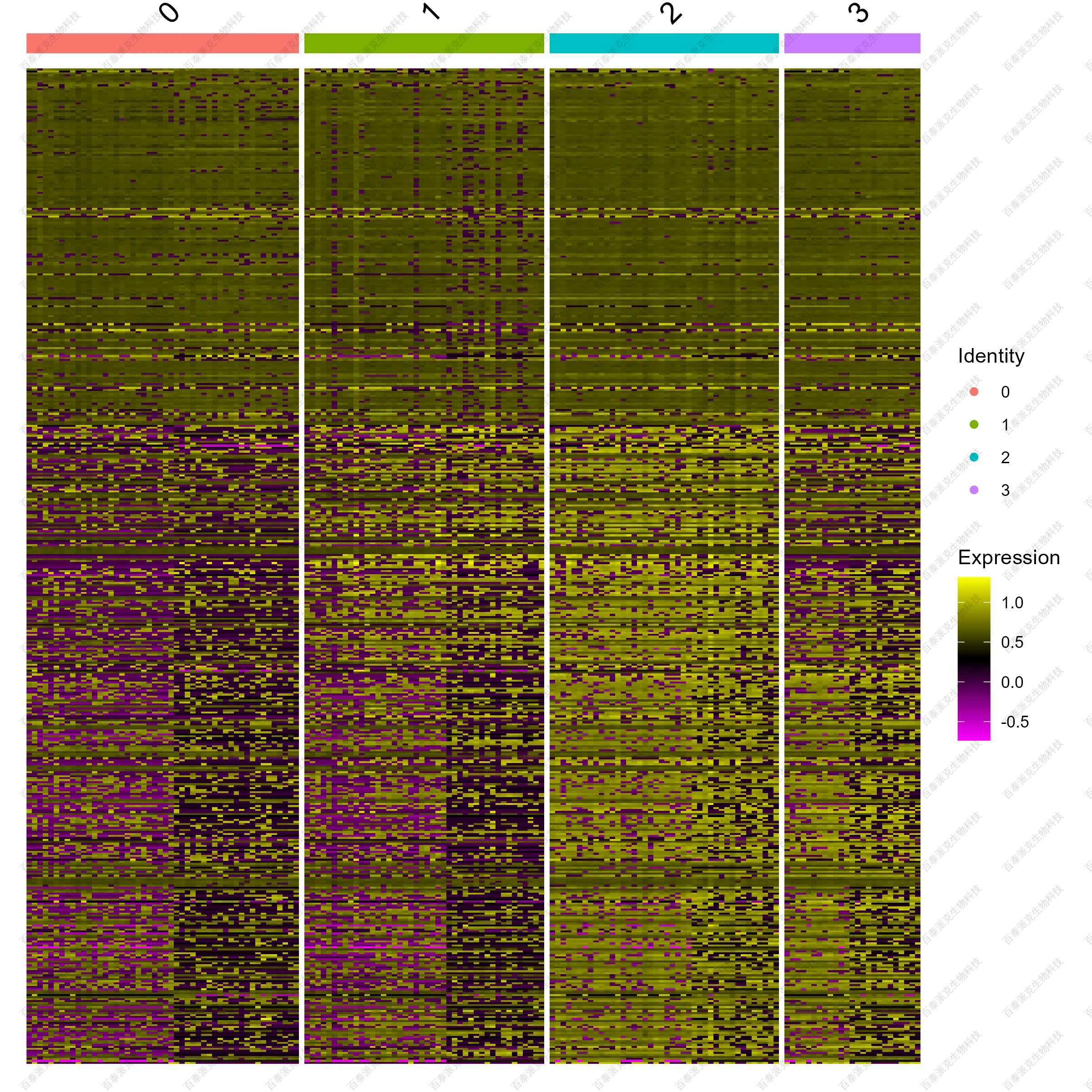
图10. 单细胞蛋白丰度聚类热图
注:横向表示各蛋白,纵向表示各个细胞,热图上方用不同的颜色对Cluster进行注释;热图颜色变化的深浅表示蛋白丰度的高低。
6、差异表达分析
我们利用差异倍率FC (Fold Change) 和t-test的显著性水平p_value来筛选差异蛋白,筛选标准为:|log2FC|>=1且p_value<0.05。
如提供多样本,亦可针对样本间,或样本分组间,进行差异分析。
(1)差异结果汇总
默认将不同Cluster与其他Cluster分别进行比较,根据差异统计结果,我们将筛选的差异蛋白分为上调表达和下调表达。各差异结果如下:

表7. 差异结果统计表
注:1)Compare:比较组;2)Up:上调表达的差异蛋白数;3)Down:下调表达的差异蛋白数。
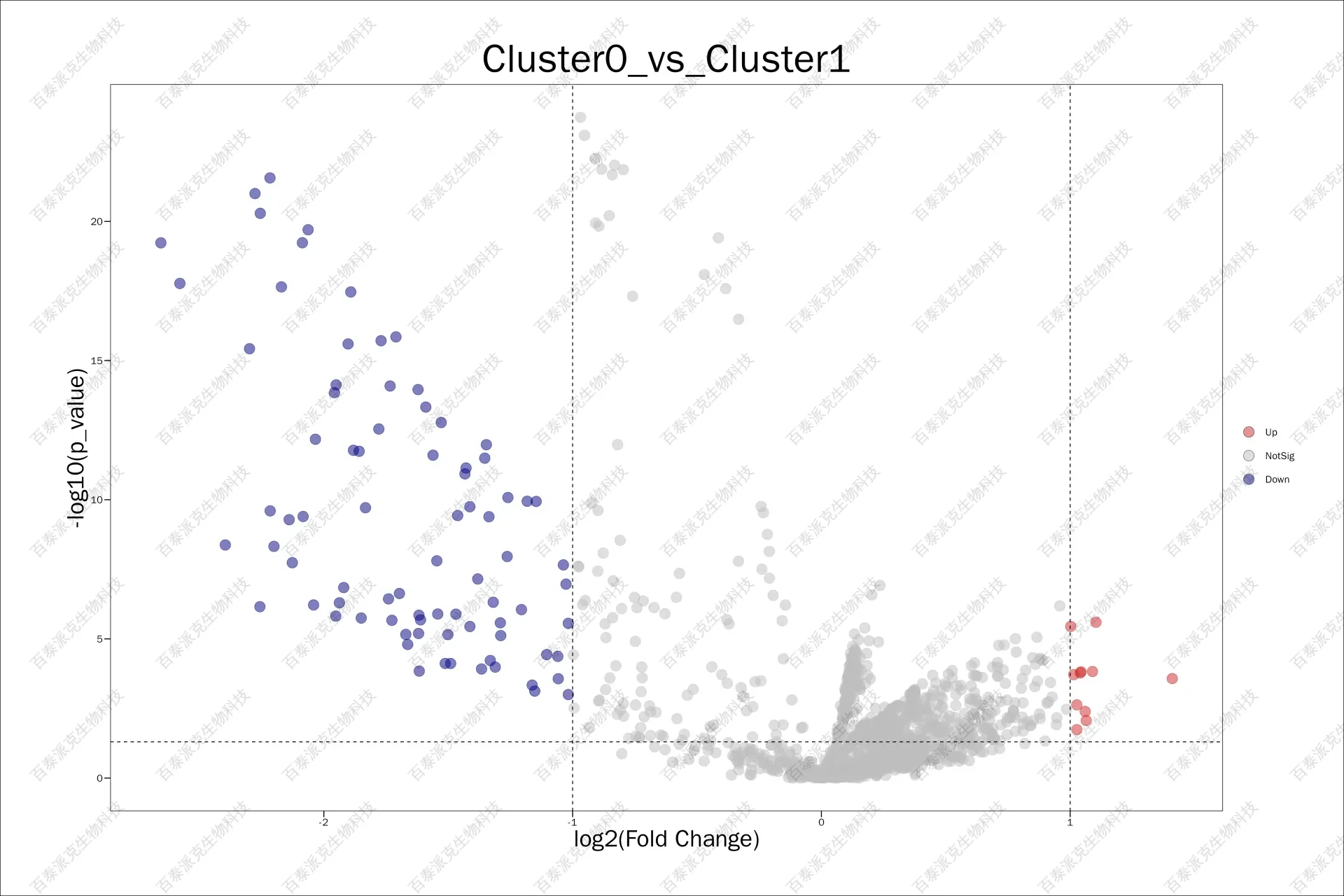
图11. 差异分析火山图
注:横坐标表示log2(Fold Change) ,纵坐标表示差异显著性。
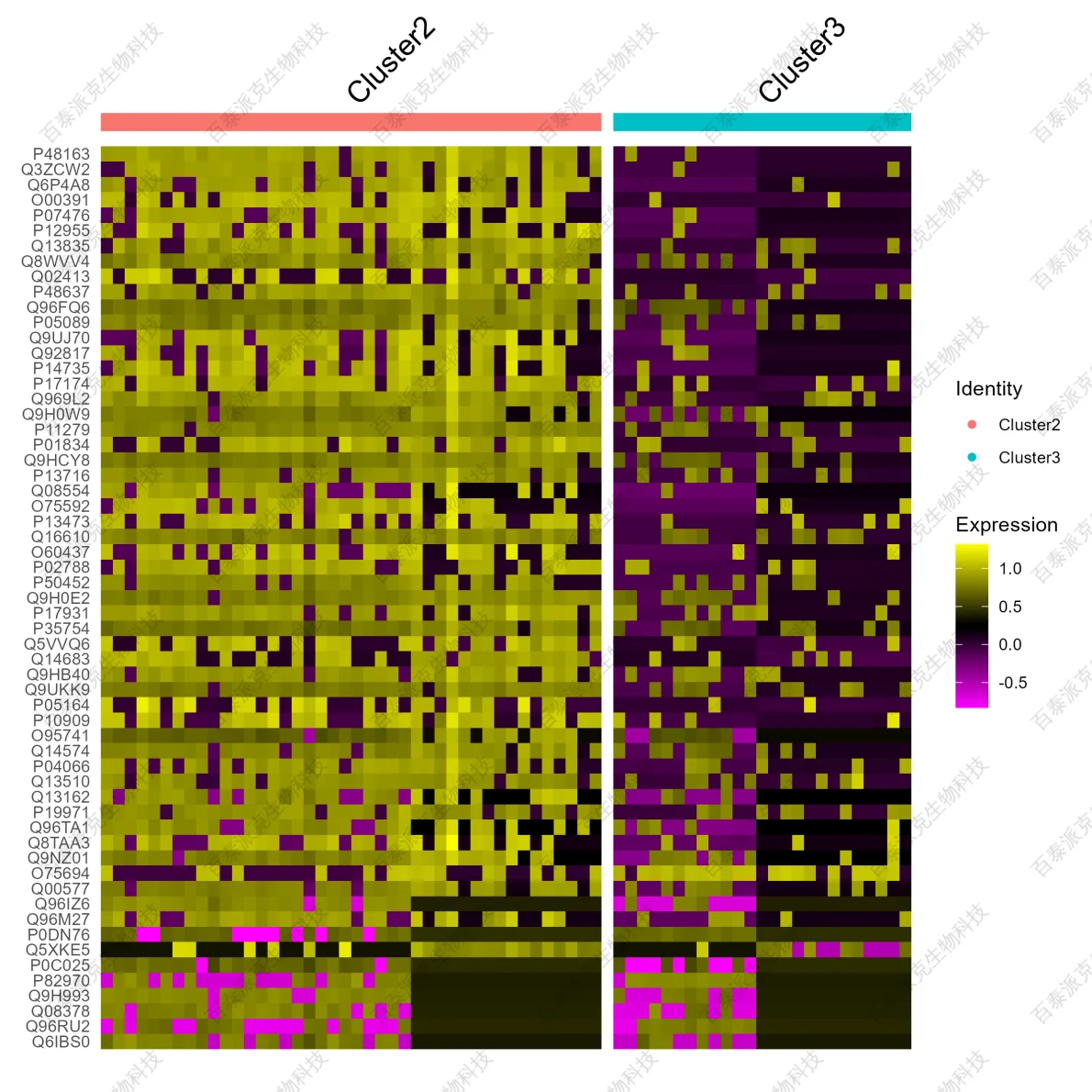
图12. 差异蛋白聚类热图
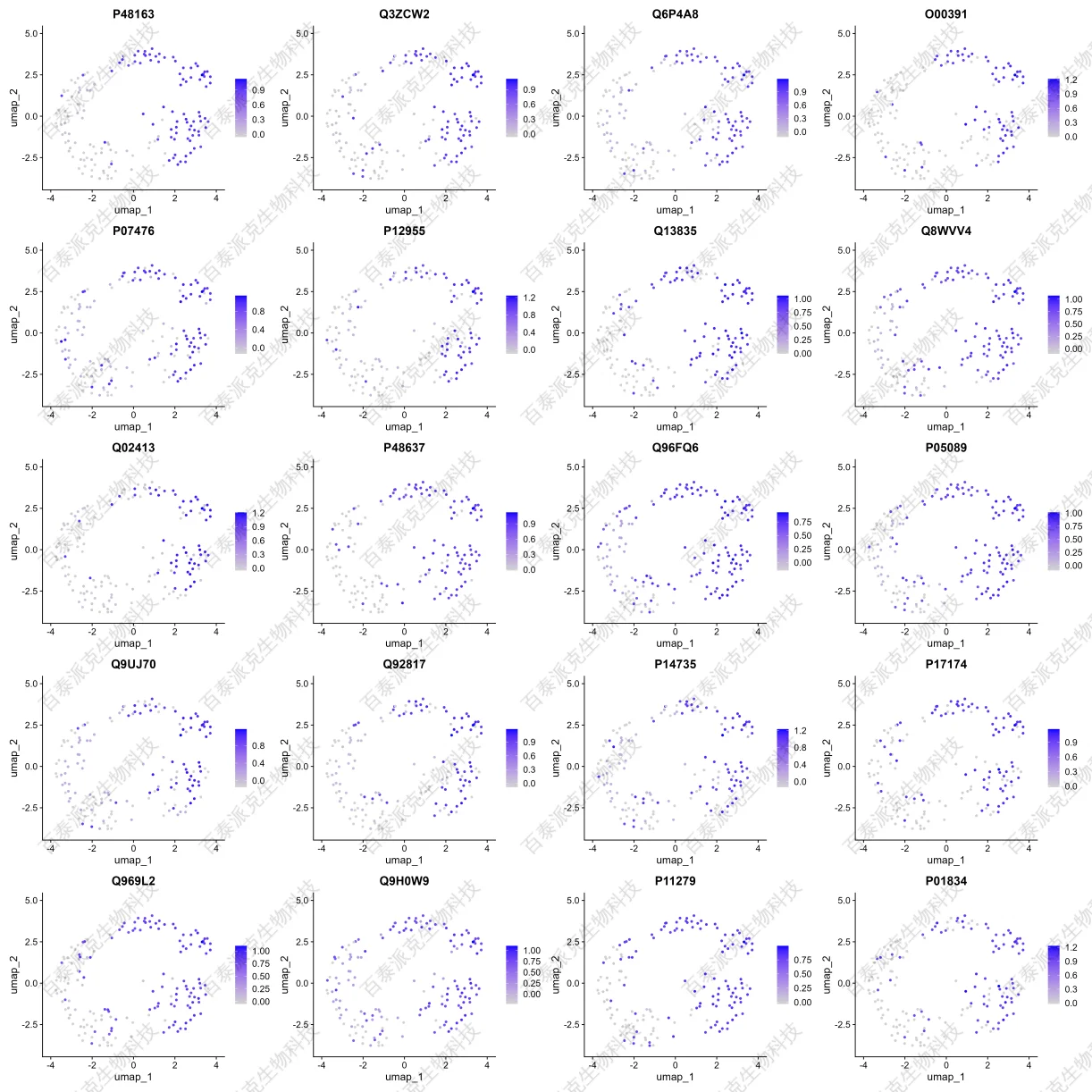
图13. Top20(p_value)蛋白丰度/降维分布图
7、功能富集分析
Gene Ontology(简称 GO, http://www.geneontology.org/)是基因功能国际标准分类体系。作为基因本体联合会(Gene Onotology Consortium)所建立的数据库,它旨在建立一个适用于各种物种的,对基因和蛋白质功能进行限定和描述的,并能随着研究不断更新。GO分为分子功能(Molecular Function,描述在分子水平上基因产物的活性元件)、生物过程(Biological Process,表示一个分子活动事件从起始到终止的过程,包括细胞、组织、器官和物种的功能整合)、和细胞组成(Cellular Component,表示细胞或其所处外界环境)三个部分。
KEGG(Kyoto Encyclopedia of Genes and Genomes)是系统分析基因功能、基因组信息的数据库,包括七大类别:Metabolism,Genetic Information Processing,Environmental Information Processing,Cellular Processes,Organismal Systems,Human Diseases,Drug Development。其中KEGG Pathway数据库是最重要也是最常用的子数据库。作为有关Pathway的主要公共数据库,KEGG提供的整合代谢途径查询,包括碳水化合物、核苷、氨基酸等的代谢及有机物的生物降解,不仅涉及了所有可能的代谢途径,而且还对催化各步反应的酶进行了全面的注解,包含有氨基酸序列、PDB库的链接等等,是进行生物体内代谢分析、代谢网络研究的强有力工具。
基于GO和KEGG Pathway数据库的富集分析,常利用超几何分布算法进行,主要是根据目标基因集合与背景相比,分析目标基因集合与哪些生物学功能/代谢通路显著相关。在蛋白质组分析中,我们以蛋白为目标基因集进行富集分析,以期阐明实验中样本差异在基因功能上的体现。
(1)GO富集分析
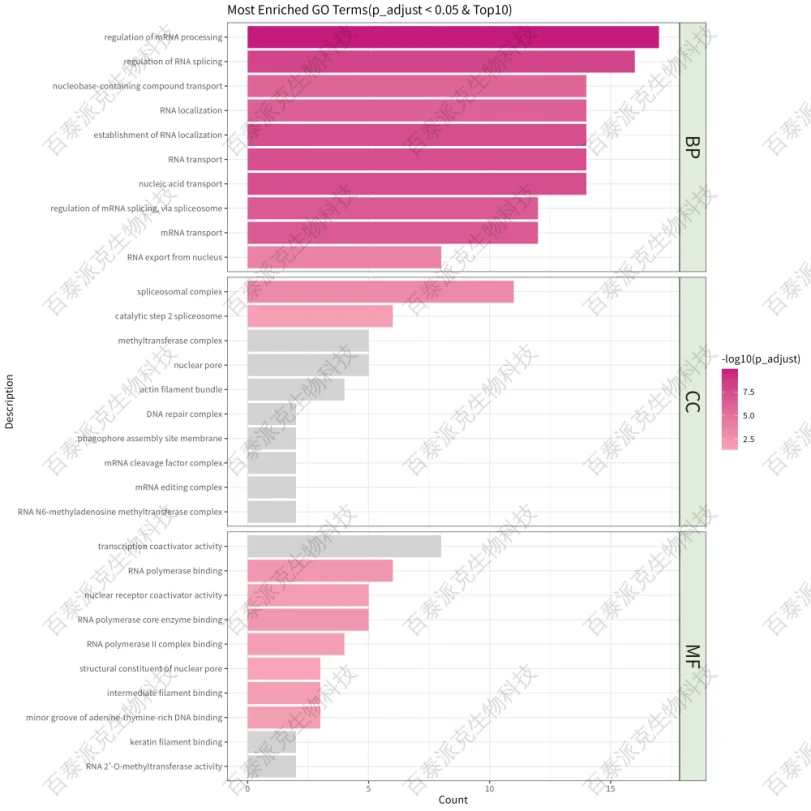
图14. GO富集柱状图
注:横坐标表示GO Term上注释到的基因数目,纵坐标表示BP、CC、MF按照p_adjust筛选Top10的GO Term;颜色深浅表示富集的显著性大小,越显著越红;灰色的柱子表示p_adjust ≥0.05的GO Term。
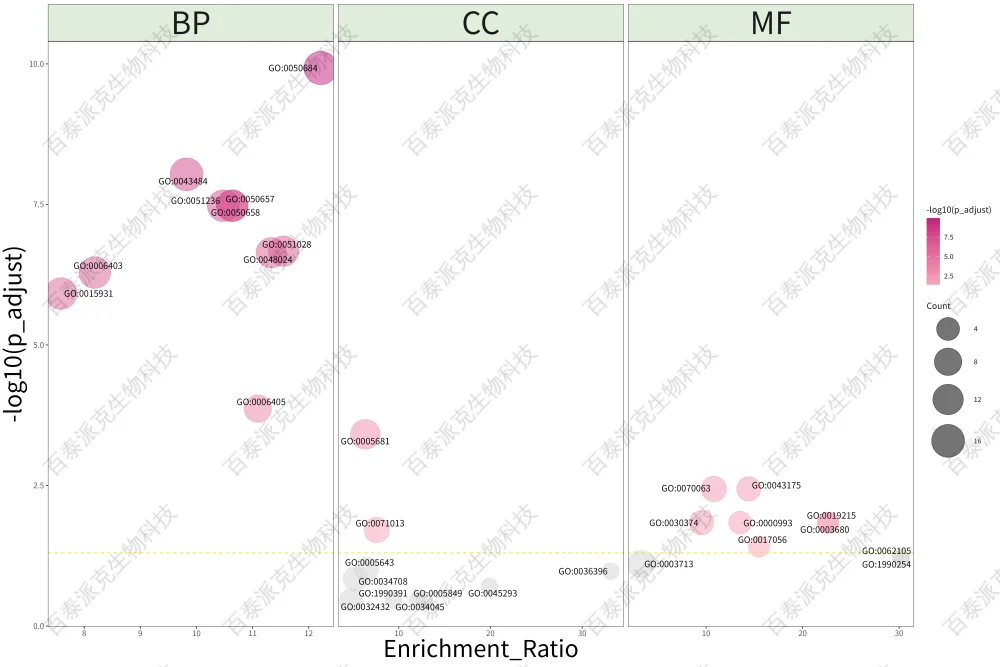
图15. GO富集泡泡图
注:横坐标表示GO Term的Enrichment Ratio,纵坐标表示GO Term的-log10(p_adjust),值越大越显著;颜色深浅表示富集的显著性大小,越显著越红;横向黄色虚线以下灰色的圆圈表示p_adjust ≥0.05的GO Term。
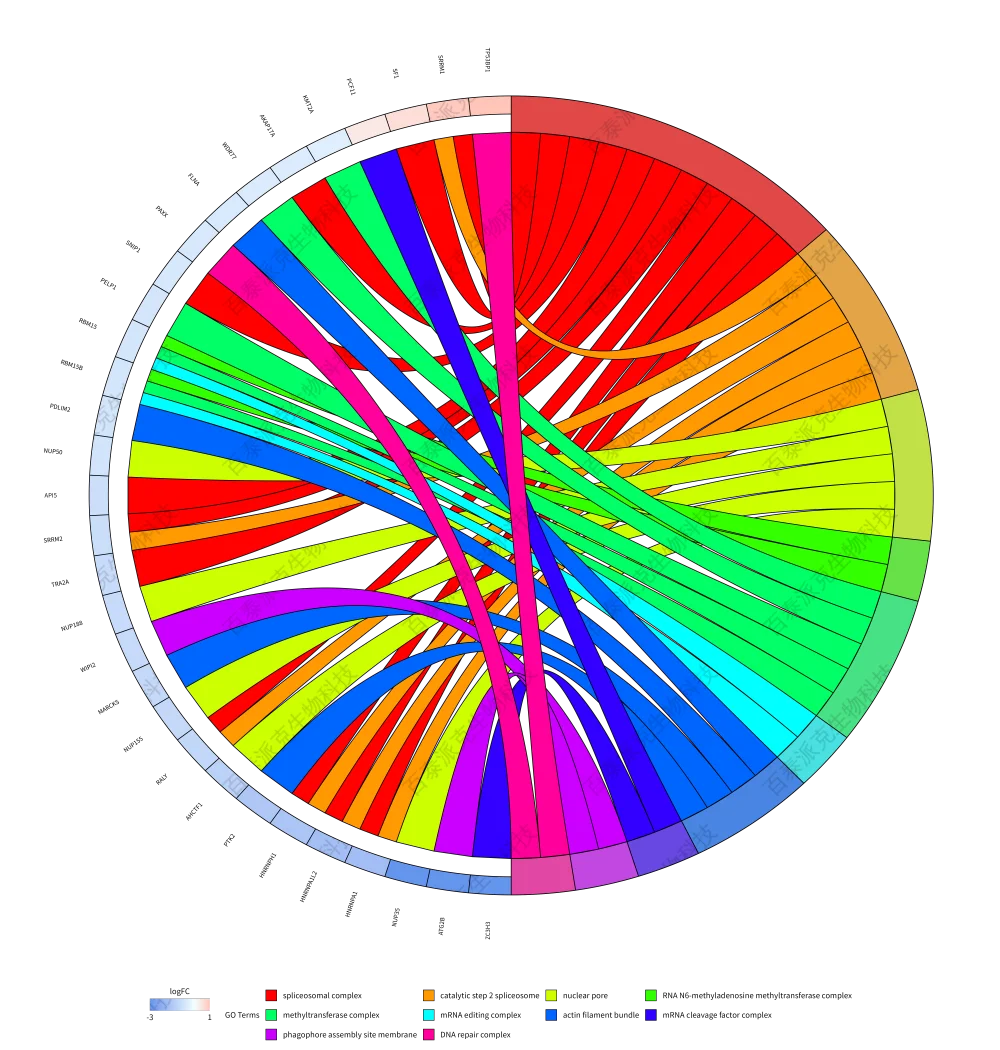
图16. GO富集弦图
注:BP、CC、MF按照p_adjust筛选Top10的GO Term(右侧);每个GO Term上对应的基因(左侧),基因颜色深浅由蛋白的|log2(Fold Change)|决定。
(2)KEGG Pathway富集分析
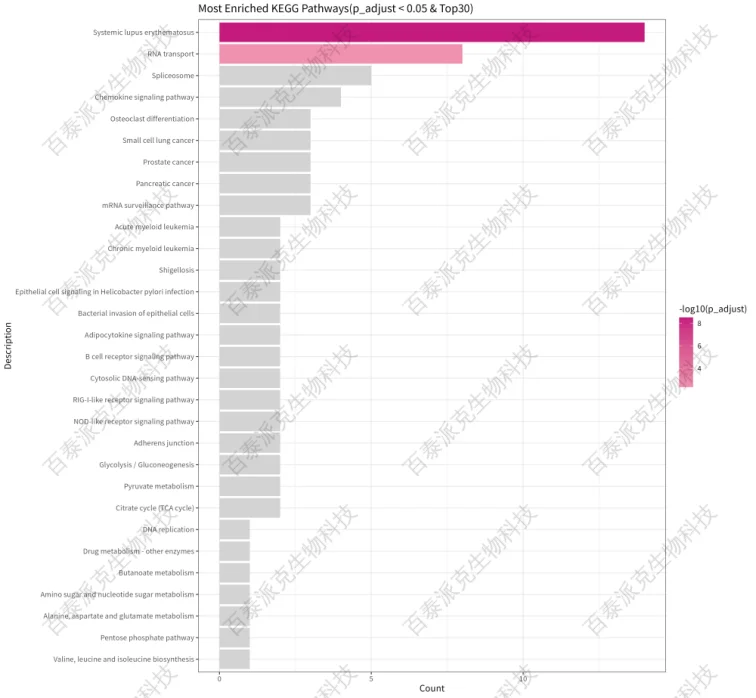
图17. KEGG Pathway富集柱状图
注:横坐标表示KEGG Pathway上注释到的基因数目,纵坐标表示按照p_adjust筛选Top30的 KEGG Pathway;颜色深浅表示富集的显著性大小,越显著越红;灰色的柱子表示p_adjust ≥0.05的KEGG Pahtway。
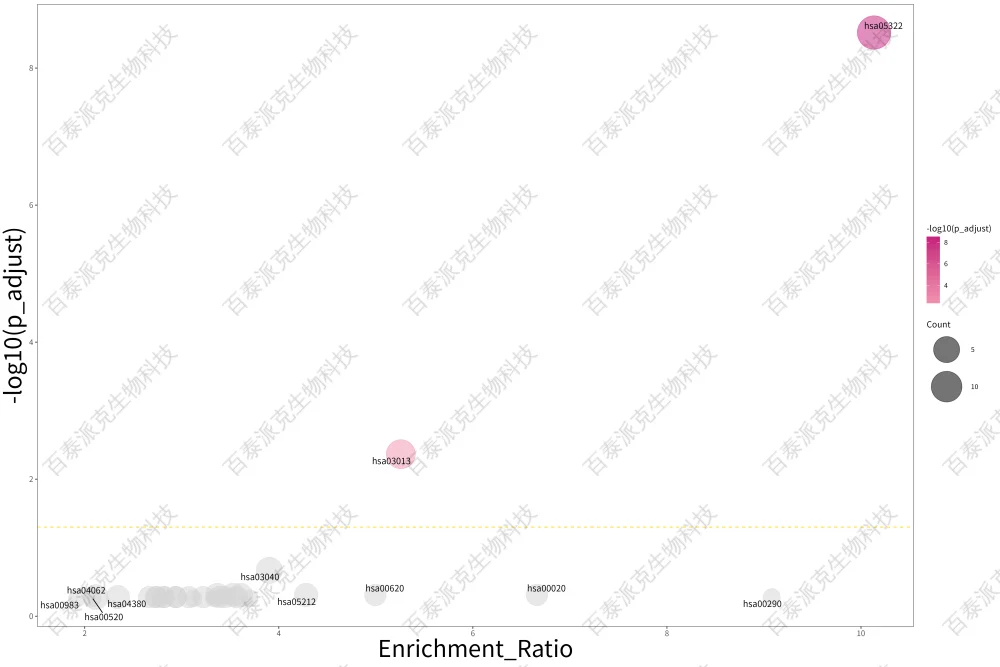
图18. KEGG富集泡泡图
注:横坐标表示KEGG Pathway的Enrichment Ratio,纵坐标表KEGG Pathway的-log10(p_adjust),值越大越显著;颜色深浅表示富集的显著性大小,越显著越红;横向黄色虚线以下灰色的圆圈表示p_adjust ≥0.05的KEGG Pahtway。
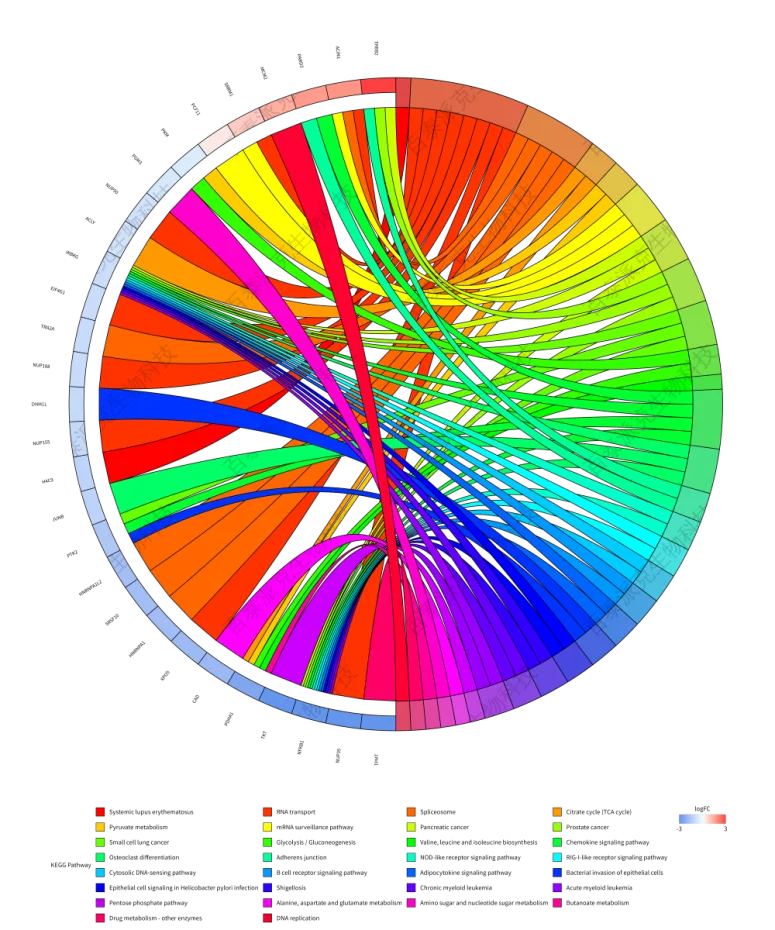
图19. KEGG富集弦图
注:按照p_adjust筛选Top30的KEGG Pathway(右侧);每个KEGG Pathway上对应的基因(左侧),基因颜色深浅由蛋白的|log2(Fold Change)|决定。
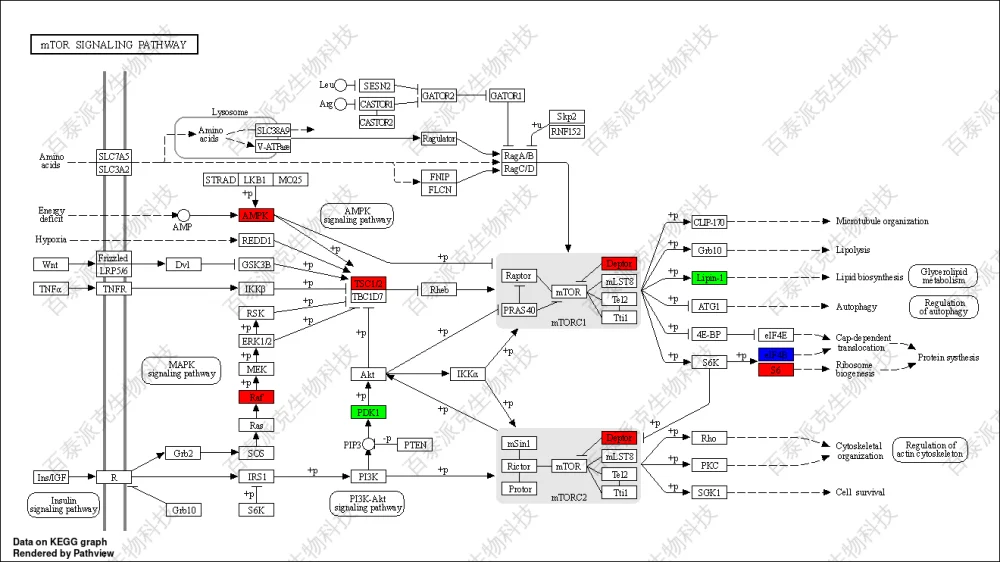
图20. KEGG通路图
注:KEGG Pathway通路图,红色的点表示基因对应的蛋白都属于上调表达,绿色表示基因对应的蛋白都属于下调表达,蓝色表示基因对应的蛋白即包含上调表达又包含下调表达。
8、高级分析
(1)细胞类型注释
单细胞类型注释是单细胞分析中的一个关键步骤,它涉及将每个细胞归类到特定的细胞类型或细胞状态。这一过程对于理解细胞异质性、细胞分化路径和细胞功能至关重要。基于现有的数据库CellMarker、PanglaoDB等能实现人、小鼠多种组织类型样本的单细胞类型注释工作。其中CellMarker数据库是专门为人和小鼠设计的,包含2w+细胞标志物,2k+细胞类型,几百种组织,数据来源10w+发表的文献,并经手动整理和注释。
我们基于现有数据库的细胞标志物信息,对项目数据进行分析,可以将每个细胞进行类型注释,甚至是亚型注释。
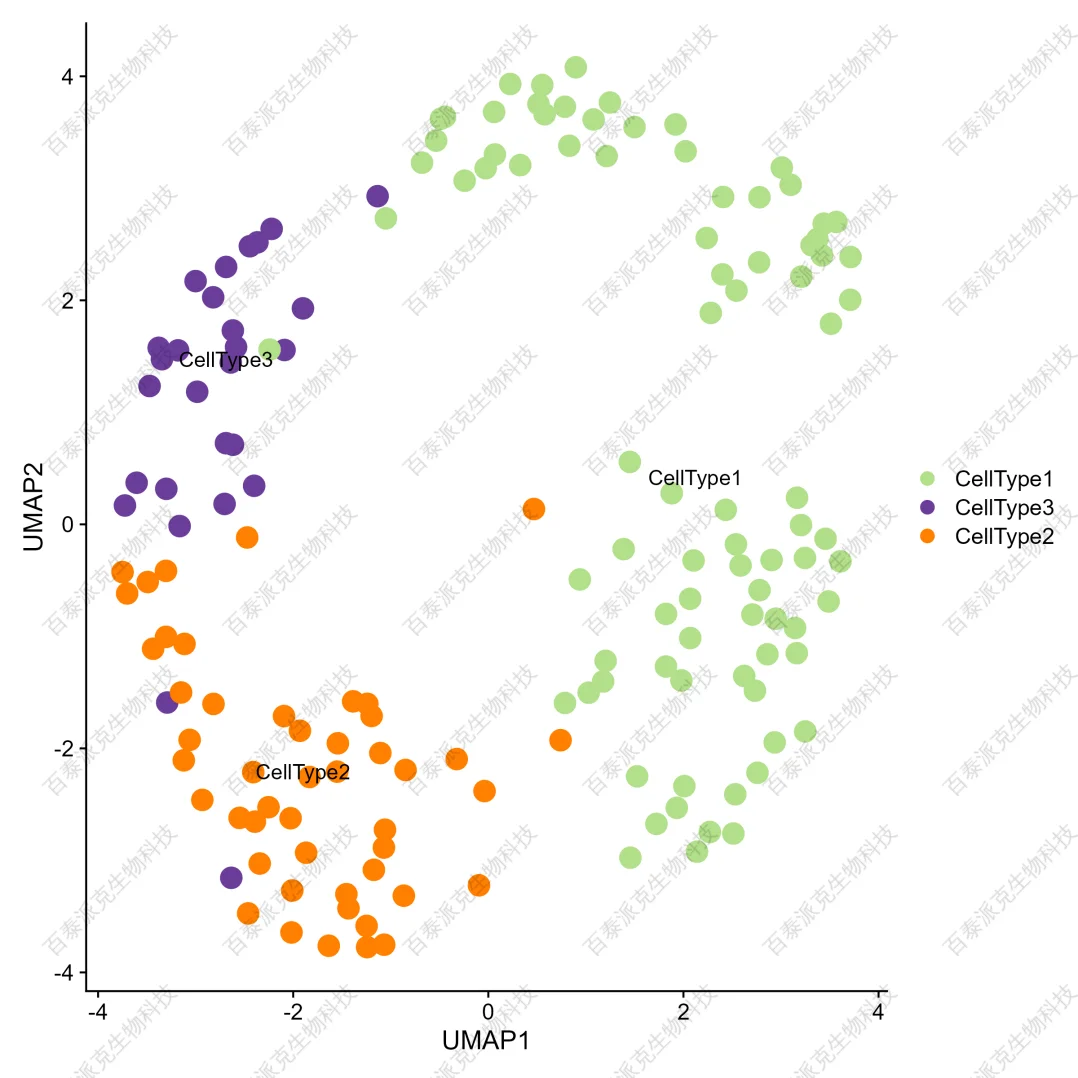
图21. 细胞注释分布图
(2)拟时间分析
单细胞拟时间分析(Pseudotime Analysis)是一种用于理解细胞在分化、生长或变化过程中所经历的状态转换的方法。它通过表达模式对单个细胞进行排序,从而模拟细胞在发育过程中的动态变化的技术,它也被称为细胞轨迹分析(cell trajectory analysis),主要用于推断发育过程中细胞的分化轨迹或细胞亚型的演化过程。这种排序技术实际上是一种在低维空间排布高维数据的降维技术。拟时间分析的应用场景拟时间分析在发育相关研究中使用频率较高,例如,在细胞分化等过程中,细胞并不是完全同步的,通过拟时间分析可以跟踪同一时间捕获的细胞间的表达变化,从而了解细胞从一种状态转换到另一种状态时所发生的调节更改的顺序。
我们利用monocle对单细胞蛋白数据进行拟时间分析。结果示例如下:
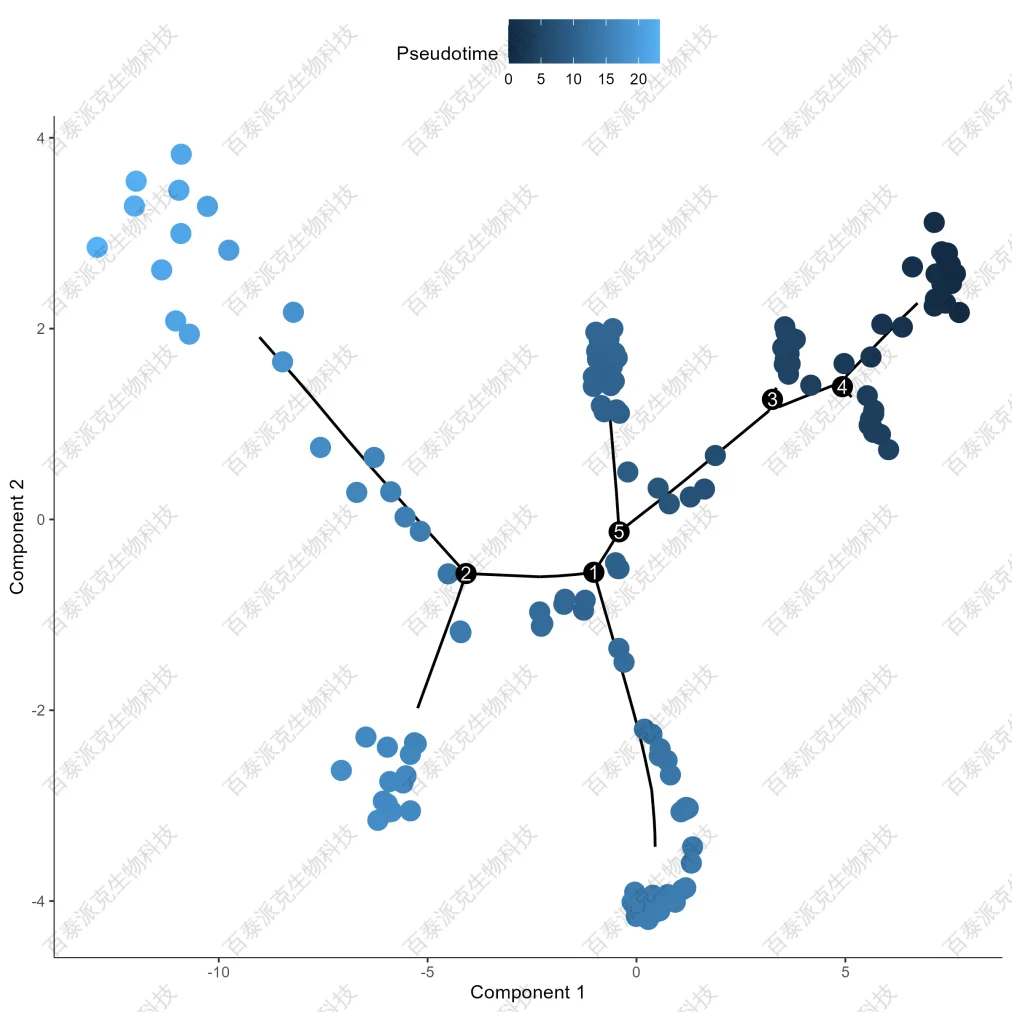
图22. Pseudotime 轨迹图
注:颜色深浅表示Pseudotime值(伪时间是一个连续的数值,代表细胞在某一发育轨迹上的相对位置。它不是真实的时间,而是一种反映细胞分化顺序的度量),不同分支表示不同演化方向,交叉位置是分支点。
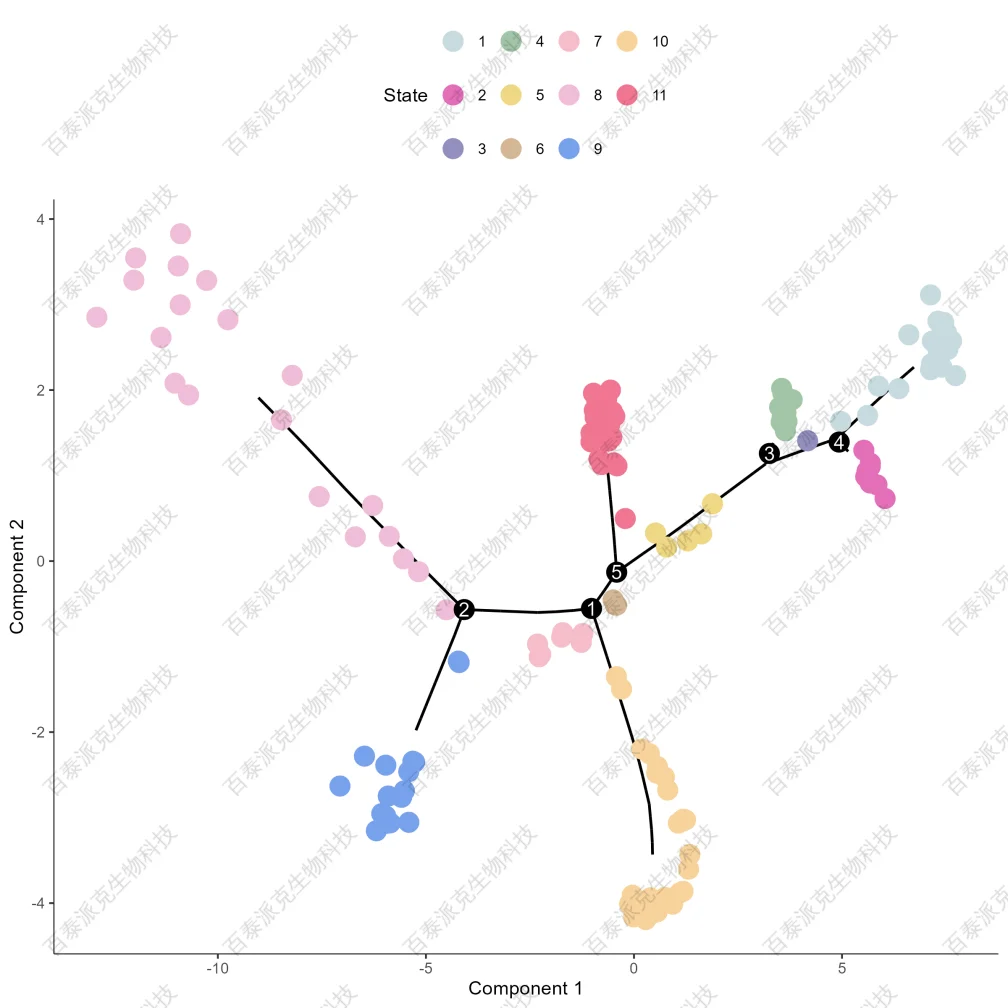
图23. Pseudotime State分布图
注:根据Pseudotime分析结果,细胞划分不同State, 及其在轨迹上的分布。
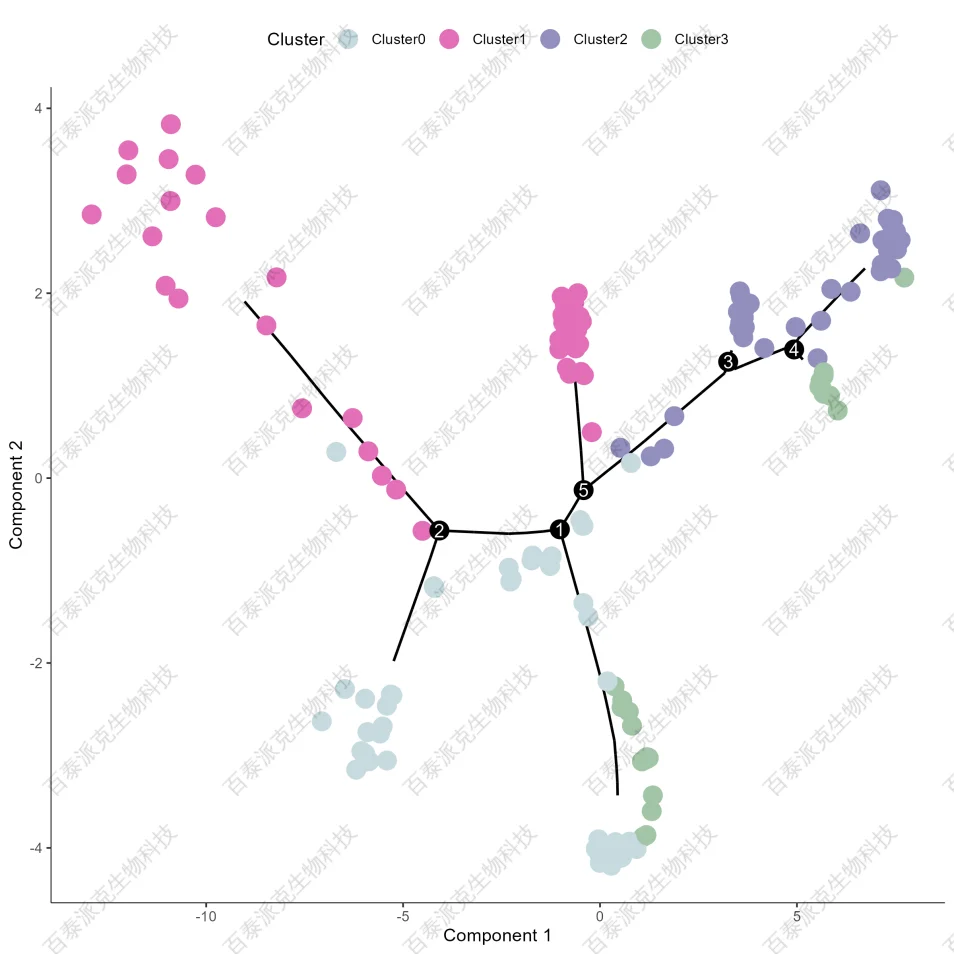
图24. Pseudotime 分群分布图
注:根据Pseudotime分析结果,结合单细胞降维分群Cluster,展示Cluster在轨迹上的分布。
单细胞蛋白组技术作为一种高分辨率的分子层面分析工具,不仅突破了传统蛋白质组学的技术瓶颈,还为揭示细胞异质性、解析细胞微环境以及探索疾病机制提供了前所未有的深度和精度。北京百泰派克生物科技有限公司作为国内领先的蛋白质组学技术服务平台,提供高质量的单细胞蛋白组检测服务。我们不仅具备规范化、自动化的样本处理流程,还在数据分析与生物信息解读方面积累了丰富的经验,确保为每位客户提供专业、精准的科研支持。我们期待与更多科研机构和企业开展深入合作,欢迎联系我们!
相关服务:
单细胞蛋白质组学分析服务
关于我们
北京百泰派克生物科技有限公司致力于为生物/制药和医疗器械行业提供质量控制检测和项目验证等专业服务。公司实验室遵循NMPA、ICH、FDA和EMA等的法规和指导原则,通过CNAS/ISO9001双重质量体系认证,建立了完备的质量体系,数据冷热/异地备份,设备定期计量/期间核查,软件审计追踪,为客户提供一体化解决方案和技术服务,支持新药研发、药物申报注册和生产放行。
1.公司采用ISO9001质量控制体系,专业提供以质谱为基础的CRO检测分析服务;
2.获国家CNAS实验室认可,为客户提供符合全球药政法规的药物质量研究服务;
3.业务范围覆盖蛋白质组学、多肽组学、代谢组学、生物药物表征、单细胞分析、单细胞质谱流式、生信云分析以及多组学生物质谱整合分析等;
4.七大质量控制检测平台,满足您一站式服务需求;
5.服务3000+企业,10000+客户的选择;
6.致力于为您提供优质的生物质谱分析服务!
技术服务一览图

